BIORREMEDIACIÓN DE SUELOS – ESTUDIO DE UN CASO EMPLEANDO BACTERIAS
TÉCNICAS BIOLÓGICAS DE DESCONTAMINACIÓN DE SUELOS
Estimulan el crecimiento de organismos en suelos contaminados, con el fin de degradarlos o extraerlos mediante la realización de sus actividades metabólicas. Se basan en la adaptación de los organismos al medio y en proporcionar condiciones adecuadas para su desarrollo (nutrientes, humedad, pH, temperatura, etc.). Se aplican a multitud de contaminantes orgánicos y a algunos contaminantes inorgánicos.
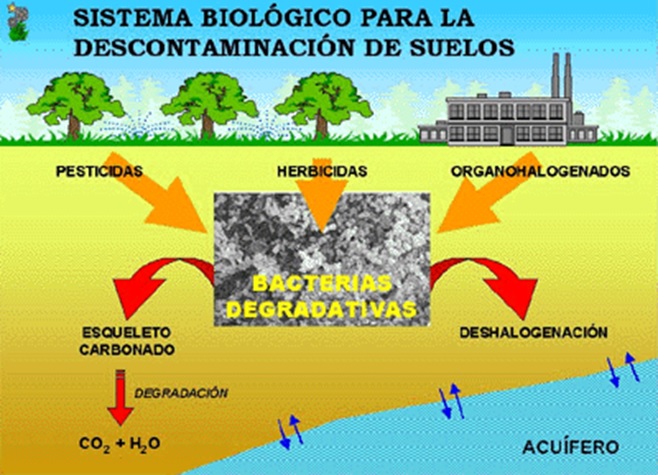
BIORREMEDIACIÓN
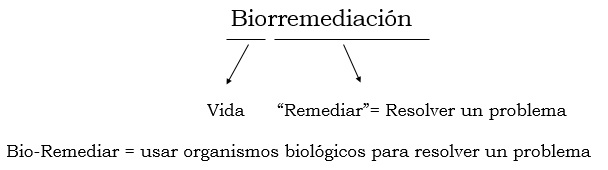
Métodos de remediación que utilizan organismos (como bacterias, plantas, hongos, etc.) o productos metabólicos para degradar contaminantes orgánicos peligrosos o convertir contaminantes inorgánicos en compuestos ambientalmente menos o no tóxicos.
Principio de la Biorremediación
La biorremediación se basa en la idea de que los organismos son capaces de tomar cosas del ambiente y usarlas para su crecimiento. En esta característica se fundamenta el principio de la biorremediación; usar organismos para que tomen sustancias contaminadas del medio ambiente y las conviertan en una forma no tóxica. Algunas bacterias, protistas, y hongos son muy buenos en la degradación de moléculas complejas.
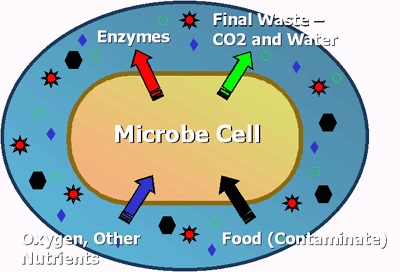
Proceso de la Biorremediación
- Los microorganismos producen enzimas que “rompen” la molécula contaminante en partes digeribles.
- El contaminante es ingerido y digerido por la célula como nutriente junto con otras fuentes de energía.
- Convertir sustancias que son peligrosas para los organismos vivos a productos inertes, de manera que solo queden desechos inofensivos de dichas sustancias.
Principales factores a tener en cuenta
Temperatura, disponibilidad de nutrientes inorgánicos (fuentes de fósforo y nitrógeno), pH, concentración de metales pesados, concentración de bacterias.
INFORMACIÓN AL RESPECTO: A continuación les comparto dos presentaciones donde se pone a disposición mayor información. (Sujeto a comentarios constructivos, que me permita mejorarlos).
Sintesis de Tecnicas de Biologicas para Descontaminacion de Suelos
Tecnica de biorremediacion Landfarming
BIORREMEDIACIÓN DEL MERCURIO
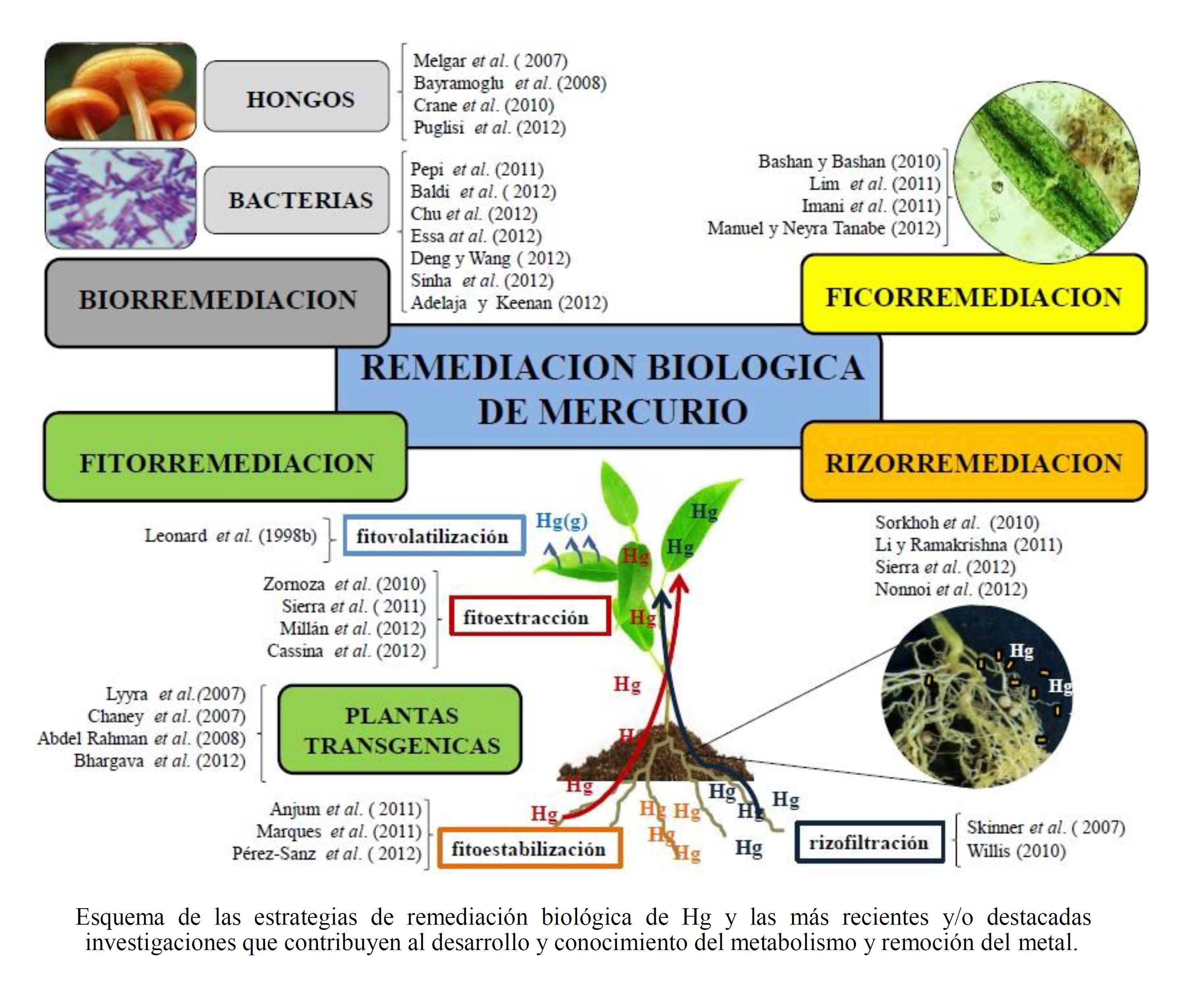
Reducción enzimática de mercurio
La presión selectiva ejercida por los ambientes contaminados con metales ha llevado al desarrollo de sistemas de resistencia microbianos, hipotéticamente, para todos los metales tóxicos, entre ellos el Hg.
El mecanismo de resistencia más ampliamente descripto para este metal se basa en la presencia de grupos de genes organizados en un único operón, denominado “operón mer”, que permite la detoxificación enzimática del metal. Estos mecanismos han sido estudiados en profundidad desde la década del ’60. Desde entonces, se publicaron una gran cantidad de trabajos en los que se describe la organización y la expresión de este operón. La estructura del operón mer varía entre las distintas especies bacterianas, encontrándose dos tipos de operones: de espectro reducido (confiere resistencia a Hg inorgánico) y de espectro amplio (confiere resistencia a Hg inorgánico y orgánico).
En conjunto, estos genes codifican para una serie de enzimas que pueden demetilar el Hg orgánico a Hg inorgánico, y reducir el Hg inorgánico a Hg(0), el cual es menos tóxico y se puede liberar al medioambiente debido a su alta volatilidad. El operón mer está constituido por genes que codifican para proteínas asociadas con varias funciones, tales como regulación, transporte y reducción. La mayoría de los operones de resistencia a Hg son inducibles y se hallan bajo un control regulatorio a nivel transcripcional, tanto positiva como negativamente.
La siguiente figura muestra un esquema de la organización general de un operón mer de amplio espectro en bacterias Gram-positivas y Gram-negativas.
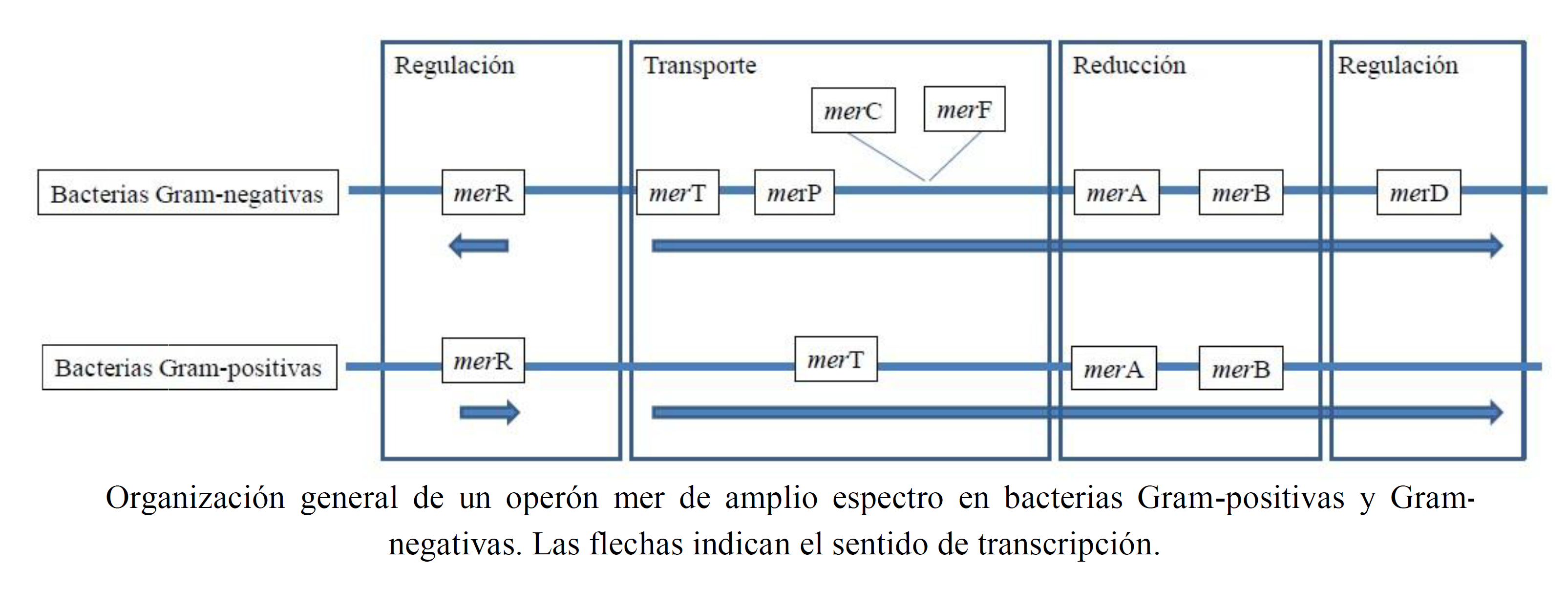
La región más estudiada del operón mer, consiste en el gen mer R, que codifica para una proteína metalo-regulatoria, la cual se une a la región del operador/promotor del operón, regulando la expresión de los genes estructurales. El gen merR se transcribe por separado y en dirección opuesta con respecto a otros genes estructurales del operón. La excepción a esta circuito regulador común del operón mer es proporcionado por el gen merR del plásmido pI258 de Staphylococcus aureus y RC607 de Bacillus sp., ambas bacterias Gram-positivas, donde éste se transcribe en la misma dirección que otros genes estructurales. La proteína Mer R activa la transcripción de los genes estructurales en presencia de Hg y la reprime en ausencia del mismo. Por otra parte, el gen mer D codifica para una proteína regulatoria secundaria que se co-transcribe junto con los genes estructurales. La proteína Mer D regula al operón mer uniéndose a la misma región del operador/promotor que Mer R y parece ser un antagonista de la función de Mer R.
El ingreso de Hg (II) hacia el interior de la célula, se considera un paso limitante de los mecanismos de resistencia a este contaminante. Los genes estructurales implicados en el transporte del Hg iónico se encuentran detrás del sitio operador/promotor. Todos los operones mer descriptos hasta el momento poseen los genes mer T y mer P, los cuales son estrictamente necesarios para la expresión completa del operón mer. La proteína Mer P se localiza en el periplasma celular y posee residuos cisteína a los cuales se une el Hg (II), luego éste se transfiere a los residuos cisteína de la proteína Mer T, localizada en la membrana celular. Finalmente, el Hg (II) es transferido a grupos -SH de la enzima mercurio reductasa codificada por el gen mer A. Mer C y Mer F son proteínas de unión a membrana y actúan como proteínas transportadoras de Hg (II), colaborando con el transporte de este ión hacia la enzima mercurio reductasa. Sin embargo, la vía de ingreso Mer P-Mer T es más eficiente que la Mer C-Mer F.
El gen mer A, codifica para la enzima mercurio reductasa, y mer B, codifica para una enzima organomercurial liasa. La proteína Mer A es una flavoproteína con dos residuos de cisteínas adicionales, que cataliza la reducción de Hg(II) a Hg(0) a expensas de NADPH. La detoxificación de Hg orgánico requiere la separación del Hg del residuo orgánico de la molécula, lo cual es producido por la enzima organomercurial liasa. El producto de la acción de esta enzima es un aducto tiolato-mercurio, el cual es sustrato luego para la enzima mercurio reductasa. Otros genes que codifican para resistencia a organomercuriales han sido identificados y designados como mer G y mer E, determinándose que se hallan implicados en el transporte hacia el interior celular.
El operón mer de bacterias Gram-negativas ha sido más estudiado en comparación con el de bacterias Gram-positivas, aunque se conoce que ambos tienen un conjunto similar de genes mer y están dispuestos en orden similar. Existen pocas diferencias entre ambos tipos de operones mer, sin embargo, el gen merB es más común en bacterias Gram-negativas que en Gram-positivas. Ambos tipos de bacterias poseen dos o más operones mer, un locus de espectro amplio y uno de espectro reducido, algunas veces localizados en el mismo plásmido. Además, el operón es altamente conservado entre estas bacterias, sugiriendo así un origen evolutivo común. Sin embargo, Freedman et al. (2012) demostraron que la expresión del operón mer en bacterias acuíficas, cuyo hábitat natural son ambientes geotermales ricos en este metal, fue independiente de la presencia de Hg(II), por lo que indicaron que la expresión regulada de mer fue probablemente una innovación posterior en ambientes donde los microorganismos se expusieron de forma intermitente a concentraciones tóxicas de Hg.
En función de lo anteriormente expuesto, se puede inferir que las bacterias reductoras de Hg y MeHg, representan una herramienta valiosa para la remediación de sitios contaminados con este metal. En la literatura se encuentran disponibles una diversidad de trabajos sobre bacterias con capacidad de reducir enzimáticamente el Hg inorgánico, que avalan esta hipótesis (Tabla siguiente).
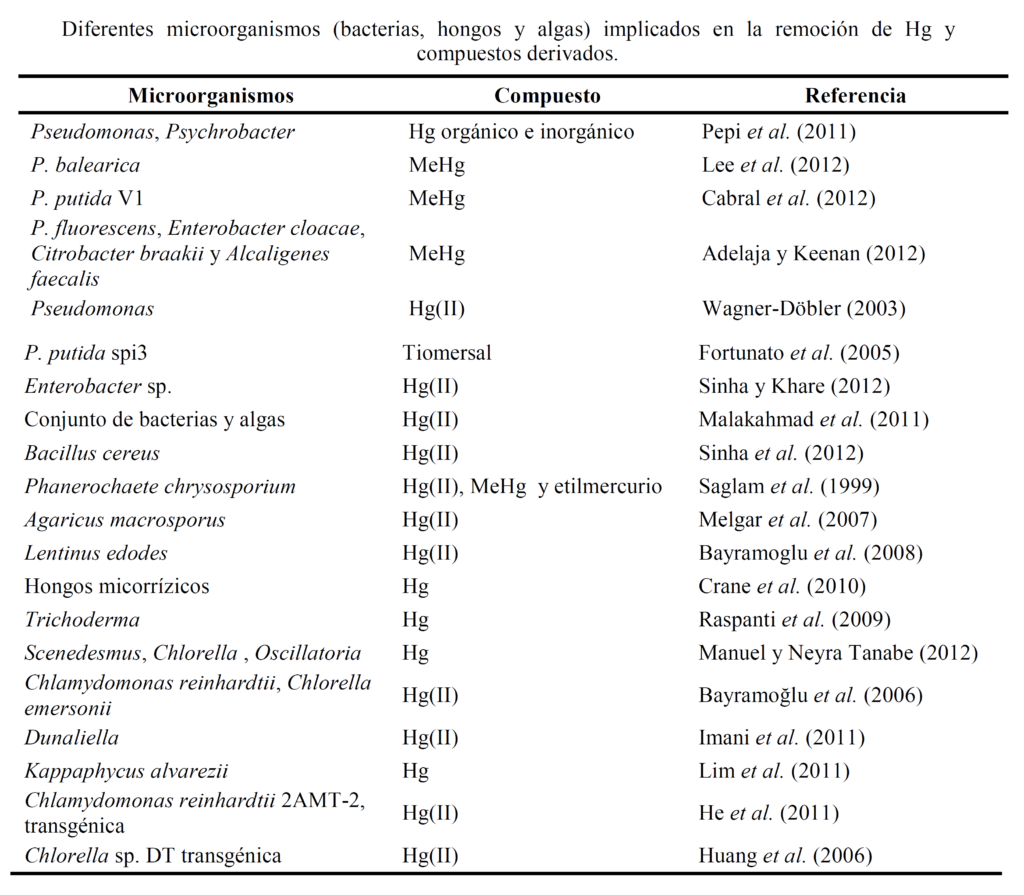
Fundamentalmente, estos trabajos se han desarrollado con el objetivo de remover Hg de medios de cultivos sintéticos líquidos con alta eficiencia, utilizando diversas especies bacterianas. En este sentido, Pepi et al. (2011) publicaron un trabajo innovador en el que utilizaron bacterias con capacidad de producir biofilms, pertenecientes a los géneros Pseudomonas y Psychrobacter, tanto libres como inmovilizadas en una matriz de piedra pómez, para volatilizar Hg orgánico e inorgánico con alta eficiencia (hasta 190 ng/mL en 5 min). Respecto de la remoción de MeHg, la mayoría de los trabajos disponibles se centran en los mecanismos enzimáticos anteriormente descriptos. Al respecto, Lee et al. (2012) describieron que Pseudomonas balearica redujo un 97% de MeHg (20 μg/L) en 3 h mientras que Cabral et al. (2012) indicaron que Pseudomonas putida V1 volatilizó un 77% de MeHg (2,5 μM), en sólo 24 h. Un aporte reciente al conocimiento de los mecanismos de detoxificación bacteriana de MeHg fue realizado por Adelaja y Keenan (2012), quienes informaron por primera vez que cepas de Pseudomonas fluorescens, Enterobacter cloacae, Citrobacter braakii y Alcaligenes faecalis utilizan MeHg como única fuente de carbono y energía.
Texto basado en la publicación “Remediación biológica de Mercurio: Recientes avances”, disponible en: http://www.ambientalex.info/revistas/vol3n23.pdf
Remediación biológica de mercurio Recientes avances